目次(まとめ)
◾️ カプランマイヤー曲線は、生存率の時間変化を可視化した図
◾️ Rの "survival" パッケージを使ってカプランマイヤー曲線を書く
◾️ 複数のカプランマイヤー曲線を書く
こんにちは、みっちゃんです。
今回の記事では「カプランマイヤー曲線を書きたいけれど方法がわからない」という方向けに、Rを使って書く方法を解説します。
カプランマイヤー曲線は、生存率の時間変化を可視化した図
ここでは、以下のようなデータをエクセルファイルで準備して、説明に使用したいと思います。
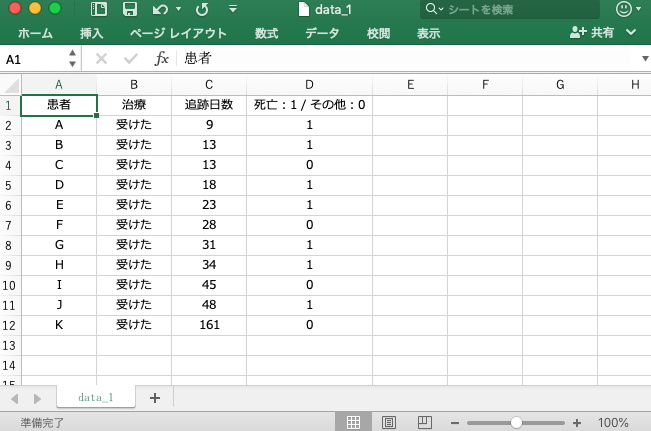
このデータは、何かの治療を受けた11人の患者さん(A〜K)について、経過を観察した時のデータです。例えば、Aさんは、治療後9日間経過を観察できましたが、死亡してしまったことを意味しており、Cさんは、治療後13日間経過を観察した結果、何らかの理由で追跡できなくなった(打ち切り)ことを示しています。
このデータを「追跡日数」ごとにまとめて、以下のように「全体の生存率」を出すことができます。
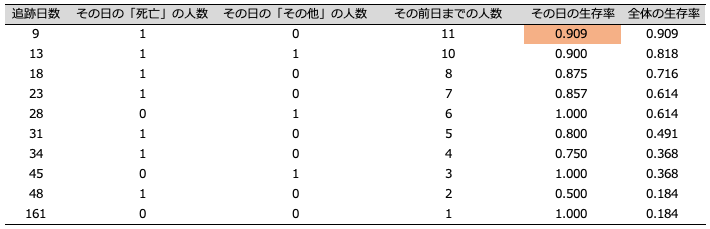
例えば、オレンジでハイライトしている数値は、"(11-1)/11 = 0.909" となっています。
「その日の生存率」をかけていくことで「全体の生存率」が算出され、この値の変化を可視化した図が「カプランマイヤー曲線」です。
Rの "survival" パッケージを使ってカプランマイヤー曲線を書く
エクセルファイルをCSV形式(コンマ区切り)でデスクトップに保存して、ファイルの文字コードを確認しておきます。
$ nkf -g ~/Desktop/data_1.csv
Shift_JISこのデータを、以下のようにRで読み込みます。
$ R -q
> data <- read.csv("~/Desktop/data_1.csv", header = T, fileEncoding="Shift_JIS")中身は以下のようになっています。
> head(data)
患者 治療 追跡日数 死亡.1...その他.0
1 A 受けた 9 1
2 B 受けた 13 1
3 C 受けた 13 0
4 D 受けた 18 1
5 E 受けた 23 1
6 F 受けた 28 0※4列目のヘッダー行の名前は「死亡:1 / その他:0」ですが、Rで取り扱えない文字が入っているので「死亡.1…その他.0」となっています。
カプランマイヤー曲線は、"survival" パッケージを使って書くことができます。
まず、以下のようにパッケージを読み込みます。
> library(survival)カプランマイヤー曲線を書くためには、以下のように実行します。
> fit <- survfit(Surv(data$追跡日数, data$死亡.1...その他.0) ~ data$治療, data)
> plot(fit, xlab = "time", ylab = "survival rate")これを実行すると、以下のような図が描けます。
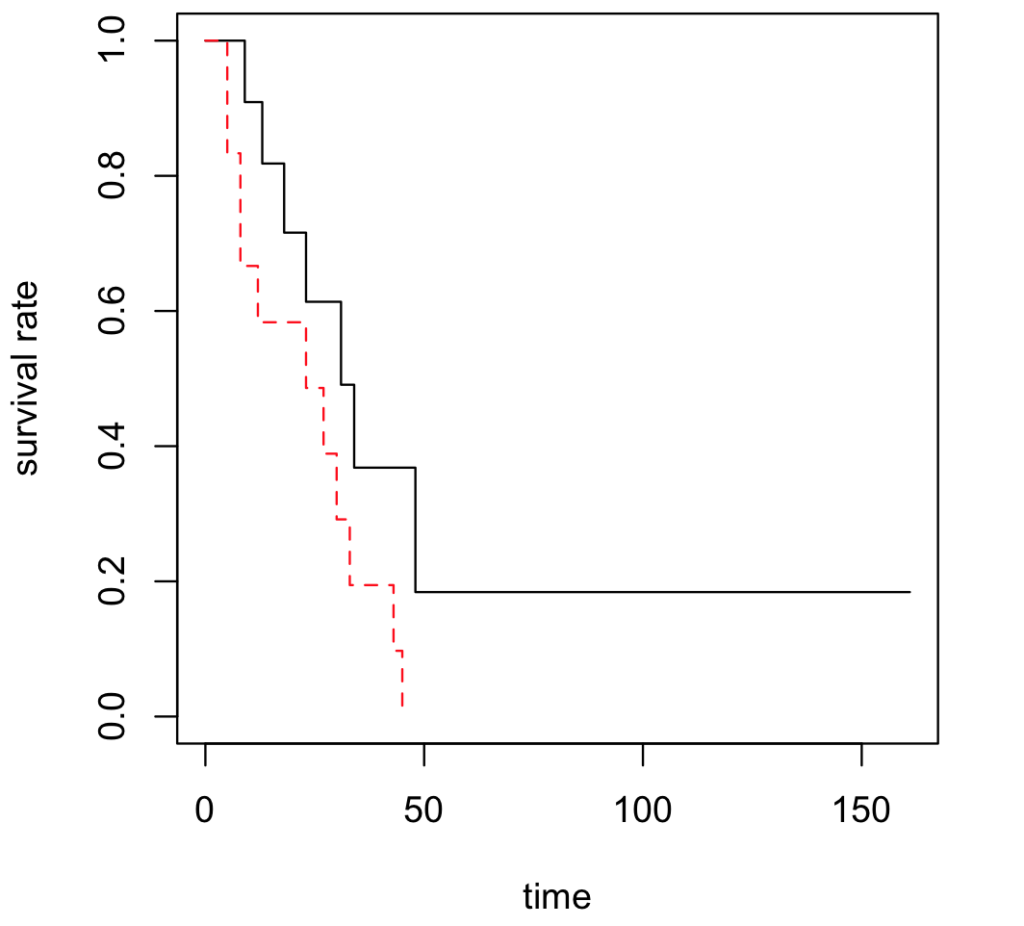
実線で描かれている曲線が「生存率」であり、破線はその信頼区間です(plot関数の中で "conf.int = FALSE" と指定すれば、信頼区間が描かれません)。
また、以下のようにカプランマイヤー曲線のサマリーを得ることができます。
> summary(fit)
Call: survfit(formula = Surv(data$追跡日数, data$死亡.1...その他.0) ~
data$治療, data = data)
time n.risk n.event survival std.err lower 95% CI upper 95% CI
9 11 1 0.909 0.0867 0.7541 1.000
13 10 1 0.818 0.1163 0.6192 1.000
18 8 1 0.716 0.1397 0.4884 1.000
23 7 1 0.614 0.1526 0.3769 0.999
31 5 1 0.491 0.1642 0.2549 0.946
34 4 1 0.368 0.1627 0.1549 0.875
48 2 1 0.184 0.1535 0.0359 0.944"survival" 列で書かれている数値が「生存率」に対応しています(上で計算していた数値と一致しています)。
複数のカプランマイヤー曲線を書く
上に示したデータでは、治療を受けた患者さんのデータだけを見ていましたが、実際には、治療を受けていない患者さんと比較したい場面が多くあります。
そこで、以下のようなエクセルファイルを取り扱います。
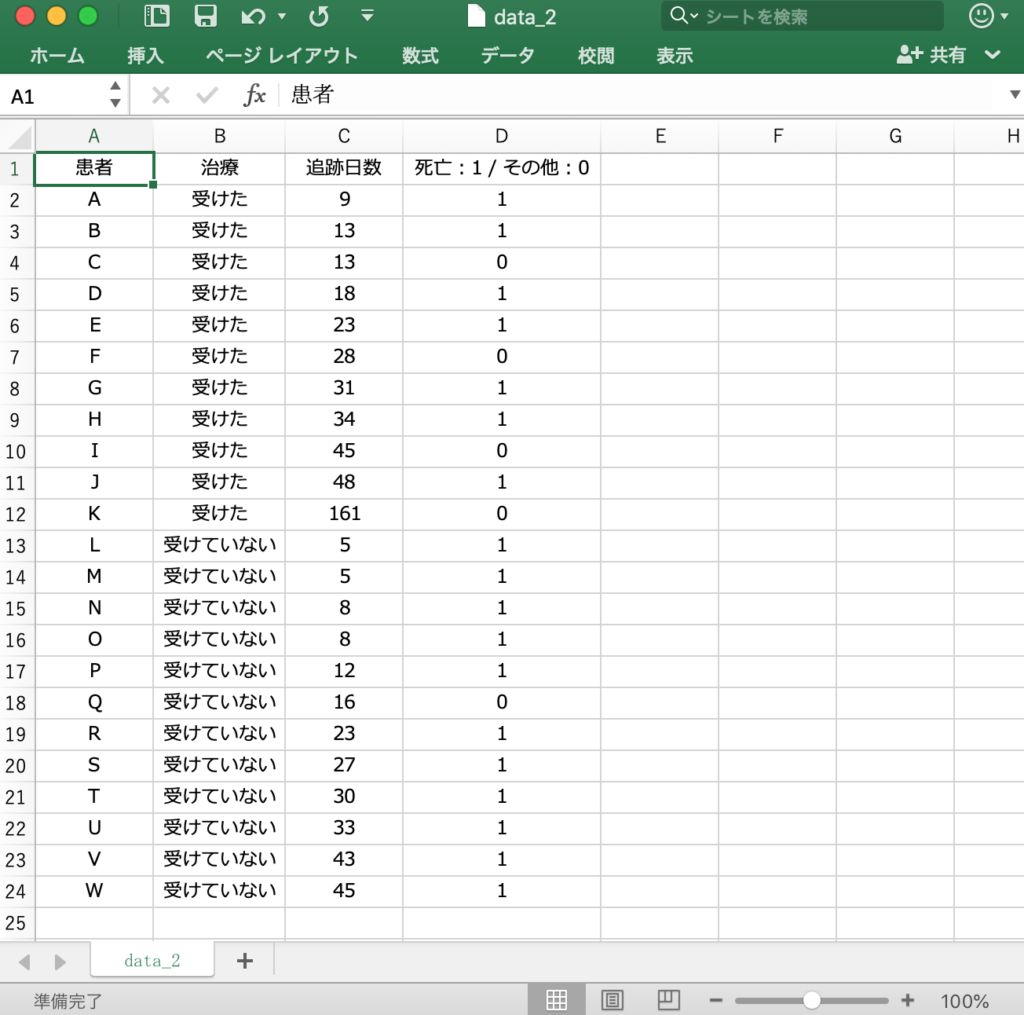
カプランマイヤー曲線の書き方は、上と同じです。
> data <- read.csv("~/Desktop/data_2.csv", header = T, fileEncoding="Shift_JIS")
# > library(survival)
> fit <- survfit(Surv(data$追跡日数, data$死亡.1...その他.0) ~ data$治療, data)
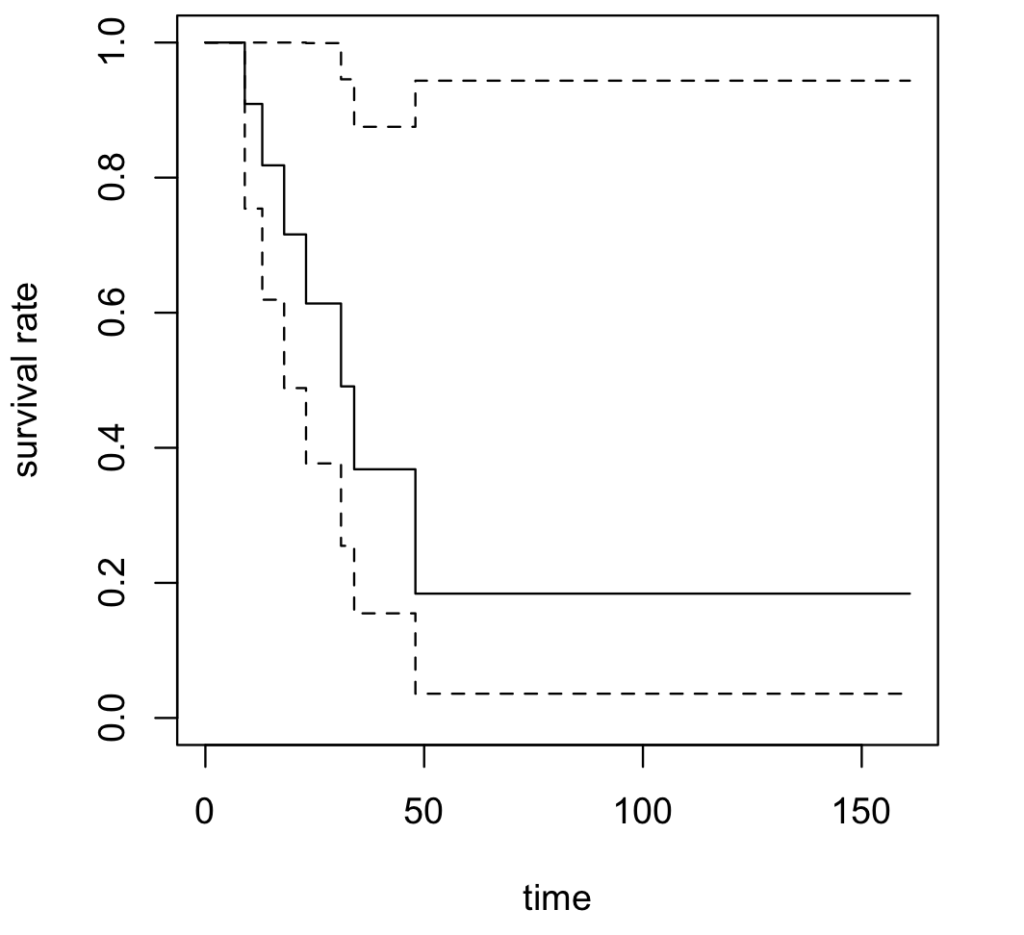
> plot(fit, xlab = "time", ylab = "survival rate", lty = 1:2, col = 1:2)"lty" は、線のタイプを指定するためのオプション、"col" は、線の色を指定するためのオプションです。