目次(まとめ)
◾️ 次元圧縮とは「高い次元」で表現されたデータを「低い次元」で表現すること
◾️ 「UMAP」は、「主成分分析」や「t-SNE」に並ぶ次元圧縮法の1つ
◾️ 関連記事
こんにちは、みっちゃんです。
今回の記事では、「高い次元」で表現されたデータを「低い次元」で表現するための手法の1つである、UMAP(Uniform Manifold Approximation and Projection)について、Rでの実行方法について紹介します。
次元圧縮とは「高い次元」で表現されたデータを「低い次元」で表現すること
ここでは、説明のために、Rにあらかじめ準備されている車の性能データ(mtcars)を使用します。
したがって、手持ちのファイルから読み込みを行う必要がありません。
以下のように実行することで、"mtcars" の一部を取り出して、データの外観を知ることができます。
> head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1ここでは便宜上、"mtcars" のスケールのばらつきを解消するため、以下のように実行して、"data" という変数に "mtcars" を読み込みます。
> data <- scale(mtcars)データは、以下のようになっています。
> head(data)
mpg cyl disp hp drat
Mazda RX4 0.1508848 -0.1049878 -0.57061982 -0.5350928 0.5675137
Mazda RX4 Wag 0.1508848 -0.1049878 -0.57061982 -0.5350928 0.5675137
Datsun 710 0.4495434 -1.2248578 -0.99018209 -0.7830405 0.4739996
Hornet 4 Drive 0.2172534 -0.1049878 0.22009369 -0.5350928 -0.9661175
Hornet Sportabout -0.2307345 1.0148821 1.04308123 0.4129422 -0.8351978
Valiant -0.3302874 -0.1049878 -0.04616698 -0.6080186 -1.5646078
:
このように、"data" は、さまざまな車の性能(例えば、mpg, cyl, dispなど)を示すデータになっています。
次元圧縮の手法は、一般的に、"data" のように、高次元の項目からなるデータを、2次元のプロットで可視化します。
「UMAP」は、「主成分分析」や「t-SNE」に並ぶ次元圧縮法の1つ
次元圧縮および可視化は、さまざまなプログラミング言語で行うことができます。
以前の記事では、「主成分分析」や「t-SNE」をRで実行する方法と、それぞれの手法で得られる結果を比較するためのツールを紹介しました。
今回の記事では、「UMAP」をRで実行する方法を紹介します。
Rで「UMAP」を行うためには、以下のように実行します。
## インストールしていなければ実行する箇所 ##
## > install.packages("umap")
## > library(umap)
> umap_score <- umap(data)> head(umap_score$layout)
[,1] [,2]
Mazda RX4 0.7022630 -1.0665393
Mazda RX4 Wag 0.3696485 -1.4252163
Datsun 710 -0.5330838 -2.5162792
Hornet 4 Drive -1.2338251 -0.6928031
Hornet Sportabout 1.1212045 3.6401369
Valiant -1.0218902 -0.5508367可視化をするためには、以下のように実行します。
> plot(umap_score$layout[,1], umap_score$layout[,2])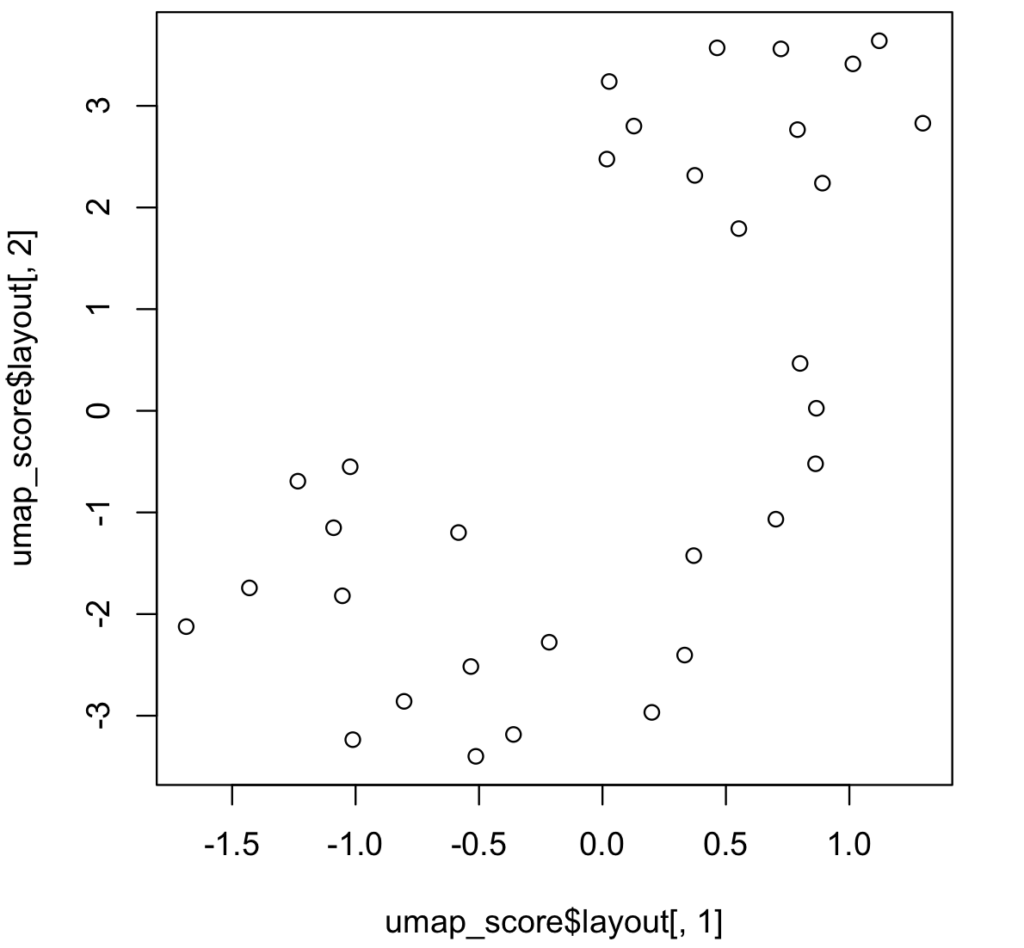
得られた結果は、以前の記事で紹介した「t-SNE」を用いた結果と異なることがわかります。